Durante quince años, el sistema nacional de salud de Dinamarca ha registrado los historiales clínicos de 6,3 millones de ciudadanos. Con todos esos datos en la mesa, un equipo de biofísicos ha utilizado técnicas de bioinformática para extraer patrones que unen enfermedades como la artritis, distintos tipos de cáncer y diabetes. Los especialistas esperan que esta hoja de ruta sea de ayuda para establecer tratamientos médicos más personalizados.
Si usted ha sufrido una angina de pecho, seguramente le gustaría saber qué otras dolencias suelen afectar a quienes han pasado por el mismo trance. Para eso, sería útil contar con algo parecido a un plano ‘callejero’ de enfermedades frecuentes con las rutas más transitadas entre ellas. De ese modo, podría anticiparse a los riesgos que le acechan en el camino.
Ese mapa existe y es danés. Los historiales de 6,2 millones de ciudadanos que han pasado por los hospitales de Dinamarca durante quince años han servido para dibujar las trayectorias entre las enfermedades más comunes de la población, tales como diabetes, enfermedad pulmonar obstructiva crónica, cáncer, artritis y enfermedad cardiovascular. El estudio, en el que han colaborado científicos de la Universidad de Copenhague, la Universidad Técnica de Dinamarca y la Universidad de Nuevo México, se publica ahora en la revista Nature Communications.
“Los daneses forman una población muy homogénea, disfrutan de unos servicios de salud unificados para todos y sus historiales clínicos están perfectamente registrados, lo que permite hacer estudios sobre la salud del país a través de la vida completa de los ciudadanos”, ha explicado el líder del trabajo, Søren Brunak, director del Centro de Análisis de Secuenciación Biológica de la Universidad Técnica de Copenhague, a los periodistas asistentes al Euroscience Open Forum 2014, que se celebra estos días en la capital danesa.
Más de mil calles entre enfermedades
Gracias a la información del Registro Nacional de Pacientes, han completado un análisis a gran escala de los más de 100 millones de datos registrados en la historia clínica electrónica de la población del país desde 1996 hasta 2010.
Con toda esta información sobre la mesa, ayudados por complejas técnicas de análisis bioinformático, han establecido 1.171 trayectorias que proporcionan un panorama completo de las enfermedades más comunes y los caminos por los que suele discurrir su historia. Cada dolencia es una parada que lleva a otras a través de las líneas de una red, como en un mapa de metro; solo que, en este caso, el viaje no se hace a través de la ciudad, sino a lo largo de la vida.
Por ejemplo, de la angina de pecho se puede pasar a la cardiopatía isquémica crónica, y de ella a la parada cardiaca. El esquema, según los autores, podría ser útil en la práctica clínica para crear tratamientos a medida; también para los epidemiólogos, que estudian la frecuencia de las enfermedades en las poblaciones y sus causas; y para diseñar políticas de salud. "Es la contribución del big data a una nueva epidemiología", dice Brunak.
Analizar grandes colecciones de datos sobre la salud en una población y extraer conclusiones de ellos es una tarea complicada, por eso, los científicos suelen centrarse en grupos específicos de enfermedades o limitarse a escalas de tiempo cortas. Sin embargo, el trabajo danés es diferente a todo lo que se había hecho hasta ahora.
Todo un país bajo la lupa de la bioinformática
Se trata del mayor análisis de este tipo, por el volumen de los datos, procedentes de 6,2 millones de personas, y el rango de tiempo que comprenden. Además, es la primera vez que se explotan los datos de la evolución de las enfermedades de todo un país.
“Podemos ver correlaciones claras sorprendentes; por ejemplo, que la gota está fuertemente ligada a las enfermedades cardiovasculares. Es una relación que los investigadores han debatido en el pasado”, ha declarado Brunak a Sinc.
No obstante, Brunak ha advertido de que estas trayectorias no explican causas ni efectos entre dolencias: “Debemos tener cuidado con la manera de interpretarlas, porque solo describen correlaciones. Para afirmar que realmente dos enfermedades tienen un vínculo habrá que buscar la explicación biológica y descartar que su relación se deba a causas intermedias que desconocemos”.
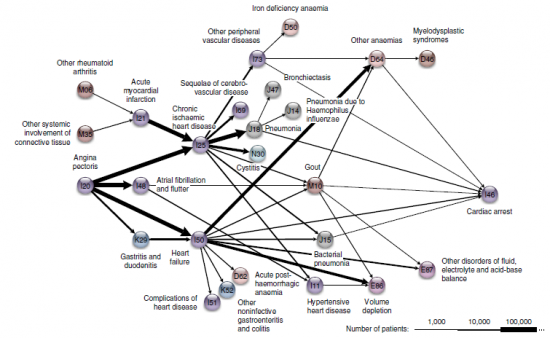
Este es el mapa de relaciones de la enfermedad cardiovascular. Se ha detectado su relación con la gota.
Lo que propone este biofísico es que los esquemas sacados de los datos clínicos se combinen con datos del genoma y el proteoma –el conjunto de proteínas del cuerpo–. “Sabemos que todos los pacientes no sufren las mismas complicaciones; depende de su ADN, de su estilo de vida, su ambiente, etc. Nuestras trayectorias estadísticas deberían servir como andamios sobre los que volcar los datos moleculares y los biomarcadores de cada paciente y construir el mapa de enfermedades por las que puede pasar”.
Predecir el siguiente achaque
Los autores del artículo creen que su trabajo puede tener aplicaciones para crear tratamientos individualizados que tengan en cuenta la totalidad del genoma y el proteoma de cada paciente. “El paso siguiente será hacer predicciones sobre los posibles riesgos de cada individuo, teniendo en cuenta los datos sobre su estilo de vida, su proteoma y su ADN”, ha explicado Brunak.
Los autores han puesto a disposición de toda la comunidad científica estos resultados cajo licencia Creative Commons 3.0.
Referencia bibliográfica:
Jensen, A. B. et al. “Temporal disease trajectories condensed from population-wide registry data covering 6.2 million patients”. Nat. Commun. 5:4022 doi: 10.1038/ncomms5022 (2014).