Hace justo 30 años, el microbiólogo Francis Mojica descubrió, junto a sus directores de tesis en la Universidad de Alicante, unas repeticiones en el ADN de una arquea de las salinas de Santa Pola. Aquella investigación daría lugar a otras que se acabarían aplicando en revolucionarias herramientas de edición genética.
La única manera de progresar es conociendo la naturaleza hasta sus más íntimos recovecos. Es uno de los motivos por los que la ciencia básica resulta esencial, aunque los que nos dedicamos a ella sintamos que nadie sabe muy bien lo que hacemos. En mi caso, he vivido en primera persona dos ocasiones en que este tipo de investigación ha tenido un impacto enorme en nuestras vidas, y precisamente estudiando los seres vivos más pequeños: los microorganismos.
El primero fue fruto de la fusión de dos líneas de investigación totalmente básicas. Por una parte, el laboratorio de Herbert Boyer en la Universidad de California –donde tuve el privilegio de disfrutar una beca Fulbright– trabajaba en una enzima de la bacteria E. coli que corta el ADN de algunos virus que la atacan. Por otra, el bioquímico Stanley Cohen era experto en plásmidos, pequeñas moléculas de ADN que viven en las bacterias como parásitos inofensivos.
La conjunción del trabajo de ambos llevó al desarrollo del ADN recombinante, que ha sido responsable de la mayor revolución en la biología de toda la historia de la ciencia, permitiendo, por ejemplo, el desarrollo de insulina humana para diabéticos o la fabricación de las vacunas de ARN para la covid.

En la arquea 'Haloferax mediterranei' de las salinas de Santa Pola (Alicante) encontramos repeticiones en tándem en la secuencia de ADN

El segundo ejemplo ha sido todavía más próximo, ya que sucedió en mi propio laboratorio de la Universidad de Alicante a principios de los años 90. Estábamos secuenciando fragmentos del genoma de arqueas –organismos procariotas como las bacterias, pero estructural y evolutivamente diferentes–, halófilas (viven en medios ricos en sal), en busca de zonas que se expresaran diferencialmente según la salinidad.
En uno de los estudios desarrollado por el, por entonces, estudiante de doctorado Francisco J. Martínez Mojica, nos centramos en Haloferax mediterranei, una arquea de las salinas de Santa Pola que yo había aislado años antes. De repente nos encontramos con regiones de hasta 1,4 kilobases de repeticiones en tándem que llamamos TREP, por tandem repeats en inglés.

Salinas de Santa Pola. / Enrique Íñiguez Rodríguez
Junto a Guadalupe Juez, codirectora de la tesis de Francis, publicamos un artículo en 1993 –justo hace ahora 30 años– que mostraba cómo la expresión de los genes cercanos a las repeticiones (posiblemente proteínas Cas) se expresaban diferente según la salinidad del medio. Pensábamos erróneamente que su papel debía estar relacionado con la resistencia a la salinidad, ya que Haloferax vive en un amplio rango de salinidades.
En realidad, no teníamos idea de cual podría ser la función de aquellas repeticiones, pero Francis no se olvidó de esta anomalía y, cuando más tarde se hizo independiente, siguió intentando dilucidar el misterio.
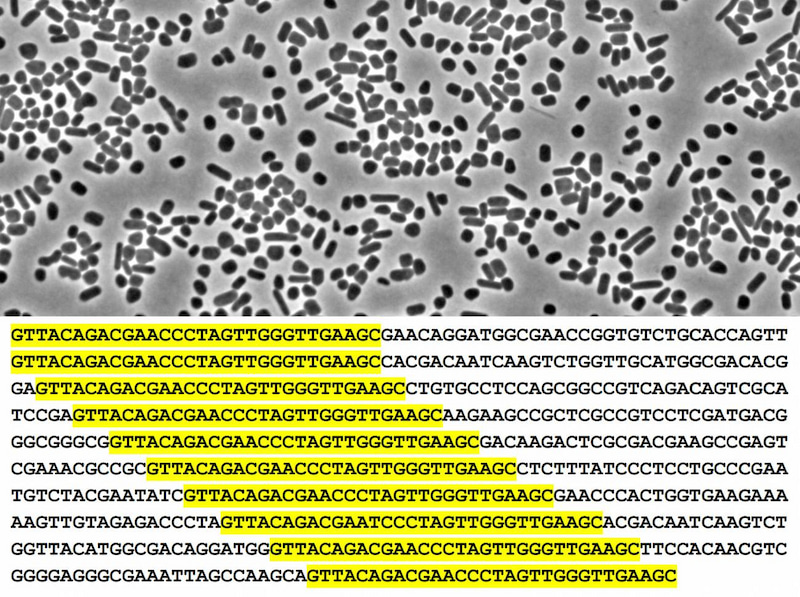
Arqueas del género 'Haloferax' y ejemplo de repeticiones en la secuencia de ADN. / Granitehead1/F. Mojica et al.
En los 'dosmiles' llegó el diluvio de genomas procarióticos y continuó buscando patrones. Muchos genomas portaban estos TREP pero nadie parecía verlos y nadie aventuraba una función. Finalmente, y junto con otros colaboradores que incluyen a mi también antiguo estudiante Jesús García Martínez de la Universidad de Alicante, se dieron cuenta de que las partes variables de las repeticiones se parecían a plásmidos o virus bacteriófagos que a veces se encontraban en los organismos a los que pertenecían las repeticiones.
Esto les llevó a plantear el nombre CRISPR (clustered regularly interspaced short palyndromic repeats) y, tras analizar bioinformáticamente las secuencias de ADN, a proponer que la función de estas repeticiones era actuar como un sistema inmunitario celular que usaban las bacterias y arqueas para defenderse de los virus. El artículo que presentaron en 2005 Francis y sus colaboradores en el Journal of Molecular Evolution explicándolo es, en mi modesta opinión, el mejor de Microbiología publicado nunca en España.

Francis Mojica y sus colaboradores propusieron en 2005 que las repeticiones actuaban como un sistema inmunitario celular que usaban las bacterias y arqueas para defenderse de los virus

Aquel descubrimiento facilitó después el desarrollo de sistemas que permiten la edición genómica de células humanas y ratones usando la proteína Cas9 de este sistema. Con esta metodología se pueden sustituir fragmentos de un cromosoma por otros sintéticos para estudiar el efecto de un cambio de secuencia o para corregir un defecto genético. Las posibilidades en terapia genética, y por ende de la medicina del futuro, son infinitas. Las investigadoras Emmanuelle Charpentier y Jennifer Doudna fueron las que desarrollaron la herramienta genética CRISPR/Cas9 y en 2020 recibieron el Premio Nobel de Química por ello.
El artículo que publicamos hace 30 años con la primera secuencia de CRISPR encontrada en una arquea ha dado pie al desarrollo posterior de esta tecnología con repercusiones globales, un buen ejemplo del poder de la investigación básica. Esta busca entender el universo en que vivimos hasta sus más mínimos detalles, y lo que hoy es ciencia básica mañana será aplicada.
Aunque existe ciencia básica buena y mala. La buena es aquella que responde a preguntas importantes y que utiliza para ello las mejores herramientas a nuestro alcance. No fue fácil desarrollarlas a principios de los años 90 en Alicante pero se lograron: las de secuenciación de ADN y la bioinformática, un campo en el que hoy sigue siendo un referente mundial.
En cualquier caso, la ciencia en España se enfrenta al reto de una mejor gestión, con órganos que detecten la calidad e incentivos reales a los buenos investigadores. No hay sistemas para captar talento internacional. A pesar de los avances, ganar un premio nobel en un entorno tan poco estimulante intelectualmente como es Alicante (incluso, a mayor escala, como la Comunidad Valenciana o España, con la posible excepción de Madrid y Barcelona) sigue siendo una utopía. A menudo se olvida que la principal riqueza de un país es el talento de sus investigadores.
Francisco E. Rodríguez Valera es profesor emérito de Microbiología en la Universidad Miguel Hernández. (A comienzos de los años 90, en la Universidad de Alicante, codirigió la tesis de Francis Mojica sobre repeticiones de ADN en tándem en arqueas).